2020年2月12日,清华大学医学院、清华大学结构生物学高精尖创新中心李海涛研究组在Cell Research期刊以长文形式报道了题为“哺乳动物ALKBH1作为局部非配对DNA中N6-mA的去甲基化酶” (Mammalian ALKBH1 serves as an N6-mA demethylase of unpairing DNA)的研究论文。该研究通过建立稳定、可靠的体外酶活检测体系,首次鉴定出ALKBH1为一类局部非配对DNA的N6-mA去甲基化酶;并首次解析了ALKBH1单体以及ALKBH1与bulged DNA的复合物结构,揭示了ALKBH1偏好局部非配对核酸底物的结构基础,为哺乳动物基因组N6-mA的调控生物学提供了全新的研究视角。

N6-mA是真核生物基因组中除5-mC之外又一种DNA甲基化修饰碱基。基因组N6-mA的水平在早期胚胎发育和癌症发生等过程中呈现出剧烈的波动,其背后的调控机制是解码这一新型修饰碱基生物学功能的关键。此前ALKBH1被报道为DNA N6-mA的去甲基化酶,但ALKBH1的生理底物一直存在巨大的争议,亟待进一步研究。ALKBH1是Fe(II)-α-酮戊二酸双加氧酶超家族中AlkB家族的成员,与5-mC去甲基化酶Tet家族存在很近的亲缘关系。在本研究中,我们首先建立了基于液相-质谱(LC-MS/MS)的定量酶活检测体系,系统地检测了ALKBH1对各类核酸底物的去甲基化酶活,发现ALKBH1偏好催化bubbled DNA而非单链DNA中N6-mA的去甲基化,且完全无法催化双链底物的去甲基化;进一步实验表明,ALKBH1可以同样高效催化多种局部非配对核酸(如bulge、R-loop、D-loop和stem loop等)中N6-mA的去甲基化,首次清晰地揭示了ALKBH1对底物二级结构的高度依赖。
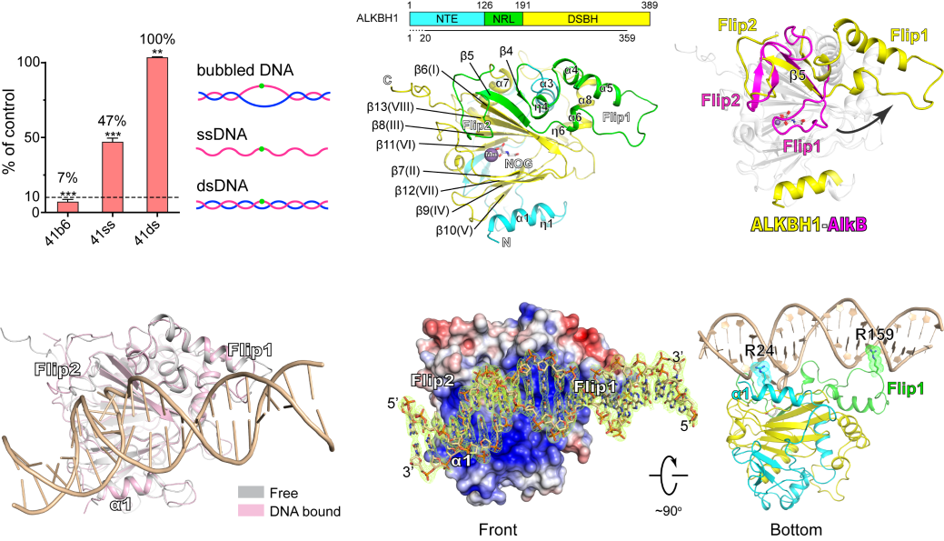
其它AlkB家族成员以及Tet家族成员均未表现出对局部非配对核酸的偏好,那么ALKBH1这种独特底物偏好的结构基础是什么呢?ALKBH1由位于C端的催化结构域(DSBH)、相邻的底物识别亚结构域(NRL)以及N端的延长区域(NTE)构成,其中NRL亚结构域又细分为Flip1和Flip2两部分。研究者首先解析了2.5 Å的ALKBH1单体结构,发现其Flip1远离酶活中心且完全外翻,与其它AlkB家族成员以及Tet2“扣回式”的Flip1形成了鲜明的反差。基于结构分析和结合实验,研究者推测外翻的Flip1很可能导致ALKBH1无法作用于双链底物,但在识别局部非配对底物的双链区中发挥了重要的作用。研究者通过引入定点交联的方式,使原本结合较弱的ALKBH1和bulge底物形成稳定、均一的复合物,并最终解析了2.4 Å的ALKBH1-bulged DNA复合物结构。ALKBH1与底物结合时其Flip1依然保持完全外翻的构象,导致其无法主动外翻底物中的N6-mA,因此完全无法催化双链底物的去甲基化。外翻的Flip1与α1位于催化中心两侧,分别与bulge左右两侧的双链区域互作。其中,α1和Flip1上的R24和R159残基分别以“Arginine finger”的形式插入双链区域的小沟中,协助将bulge上的N6-mA呈递至催化中心,凸显了局部非配对底物双链区域在底物识别过程中的关键作用,清晰地阐明了其偏好催化局部非配对核酸底物而非单链底物的原因。研究者进一步对小鼠早期胚胎发育细胞进行DIP-seq以及ssDNA-seq分析,发现N6-mA与基因组的非配对区域存在着显著的共定位,暗示着N6-mA的调控可能与真核生物染色体高级结构的动态变化密切相关,这将为后续ALKBH1以及N6-mA的功能研究提供全新的视角。
清华大学李海涛教授以及耶鲁大学Andrew Xiao教授为共同通讯作者。李海涛实验室张敏博士(生命科学中心2015级博士研究生)、医学院博士研究生杨舒敏以及耶鲁大学博士研究生Raman Nelakanti为本文共同第一作者。第二作者2018级PTN联合项目博士生赵文涛同学和代谢质谱平台的刘晓蕙老师为本工作顺利开展做出了重要贡献。
本工作得到国家自然科学基金委、科技部重点研发计划、生命科学联合中心、北京市结构生物学高精尖创新中心(清华)、北京生物结构前沿研究中心等的经费支持。本课题得到上海同步辐射光源、清华大学X-ray晶体学平台以及代谢质谱平台的支持与协助。








