2021年8月20日,清华-Science系列大讲堂“计算结构生物学”专题第三讲——“蛋白质动力学”开讲。本期嘉宾为美国希望之城贝克曼研究所计算与定量医学系教授、主任Nagarajan Vaidehi和清华大学生命科学学院副教授、博士生导师、北京结构生物学高精尖创新中心龚海鹏,清华大学生命科学学院李雪明副教授主持了本次讲座。
蛋白质动力学作为研究蛋白质静态结构和动态结构关系的桥梁, 能够揭示结构随时间变化的特征,即能够将蛋白质分子的微观运动同生物功能联系起来,从而让我们可以在原子、分子水平上理解蛋白质分子的生物功能,并揭示产生这种功能的分子机制。然而,研究蛋白质动力学并不容易,因为几乎不可能“看到”蛋白质是如何运动的。这时,计算机模拟方法就显现出了它的价值。计算机模拟通过揭示蛋白质分子复杂生物过程的原子细节,特别是那些不适合实验观测的细节,比如原子水平上的动态性质,从而极大地促进了蛋白质动力学的研究。在今天的报告中两位嘉宾便分别以《时空决定因素与变构互作对G蛋白偶联受体的调控作用》(Protein structural prediction and conformational sampling)和《蛋白质结构预测与构象抽样》(Protein structural prediction and conformational sampling)为题介绍了蛋白质动力学的研究内容。
Nagarajan Vaidehi 教授
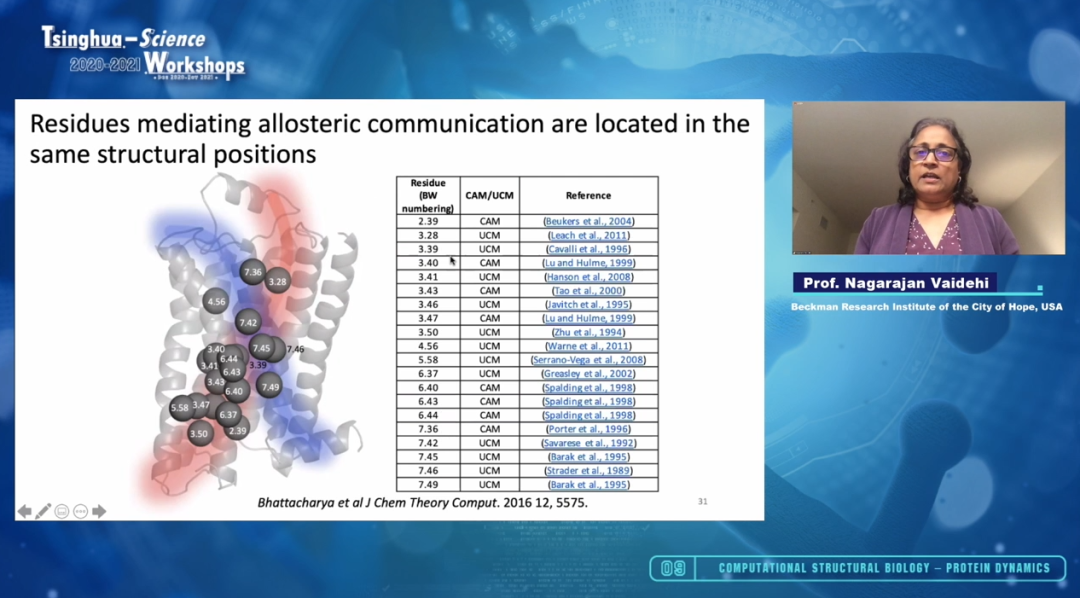
第一位嘉宾是来自希望之城计算与定量医学系主任的Nagarajan Vaidehi教授。G蛋白偶联受体(GPCR)是一大类膜蛋白受体的统称,其主要功能是通过与G蛋白相互作用将细胞外的信息传递到细胞内。G蛋白偶联受体可以识别各种配体和刺激物,包括各类激素和神经递质、前列腺素、蛋白酶、生物胺、核苷、脂类等。这些受体作为细胞内介体可以调节复杂的网络途径。Vaidehi教授在报告中介绍到,G蛋白偶联受体与G蛋白或β-抑制剂的相互作用通常是短暂且动态的,而这种相互作用在某些情况下表现出高度的选择性,在另一些情况下表现出杂乱性,在一个细胞信号事件上与一个或多个伴侣蛋白耦合。Vaidehi教授团队利用多尺度分子动力学模拟并结合FRET实验和基于细胞的分析,最后表明,时空因素在揭示GPCR对G蛋白和β-抑制剂的选择性偶联中发挥了关键作用。另外,其团队利用MD模拟还表明了部分GPCR与多个G蛋白存在混杂偶联现象。我们知道,信号蛋白的变构通讯在细胞膜信号转导中发挥着重要的作用,同时,GPCR也是药理学上重要的蛋白家族,涉及受体途径的药物数以百计,包括安定药、抗抑郁药药物以及抗高血压的药物等。Vaidehi教授还介绍到,研究表明“偏置配体”在治疗上是可取的,它将使药物设计具有最小的副作用。
龚海鹏副教授
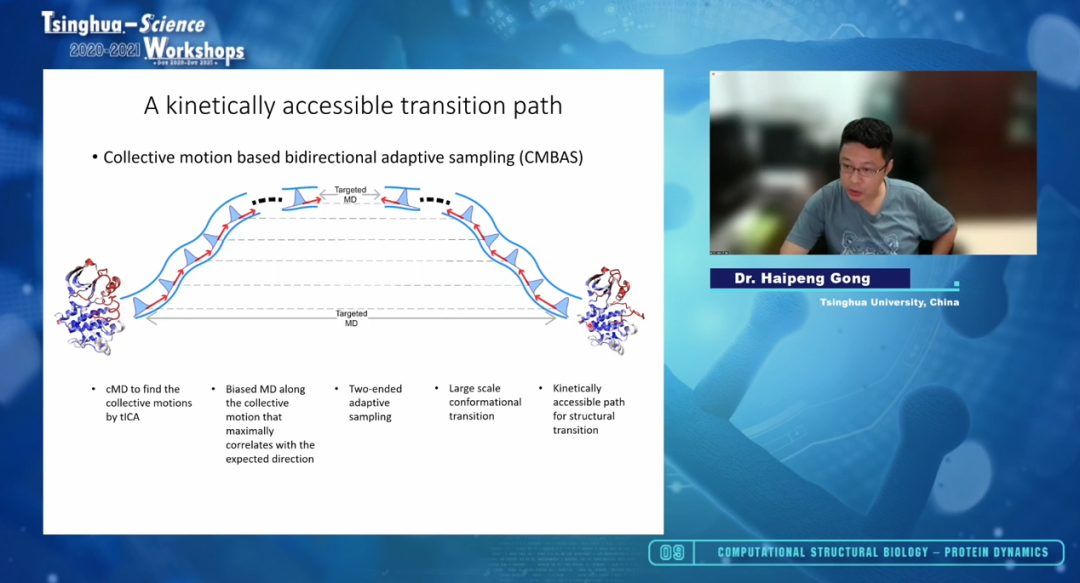
随后,在第二个报告中,龚海鹏副教授介绍了蛋白质结构预测与构象抽样的相关研究。蛋白质结构预测的方法学近年来取得了突破性进展,尤其是DeepMind公司研发的AlphaFold2,收益于人工智能的飞速发展,将预测的准确性提高到了惊人的水平。报告中,龚海鹏详细介绍了其团队在过去几年开发的两个模型——AmoebaContact和GANProDist算法。这两种算法皆属于目前创新性较高的人工智能方法,能较为准确地预测蛋白质残基间距离,并根据预测距离折叠蛋白质建立其三维结构模型。与大多数传统方法不同的是,AmoebaContact和GANProDist模型分别采用了自动神经结构搜索和对抗训练等新思路,为进一步发展和完善基于深度学习的蛋白质结构预测模型提供了新的思路。根据目前的数据,AmoebaContact在残基间距离的预测精度方面已经达到了接近RoseTTAFold的水平。报告中,龚海鹏副教授详细介绍了其团队最近开发的两种方法以及最新进展,以期未来能够解决单结构域蛋白质的精准结构预测问题。









