深度学习推动了蛋白质语言模型(pLM)的发展,使其能够设计功能性蛋白质序列并预测抗体进化,AlphaFold和ESMFold等模型的出现解决了结构预测难题。然而,pLM在预测蛋白质相互作用(PPI)方面仍存在局限,现有方法(如分别嵌入或拼接序列)未能有效捕捉关键残基接触点和上下文特异性,且受限于序列长度差异,导致其在学习"蛋白质相互作用语言"方面存在固有缺陷。

SWING是一种创新的交互语言模型(iLM),通过预先编码成对残基的生化差异信息生成蛋白质相互作用的语言表征(图1a)。该方法采用滑动窗口技术(图1b)将氨基酸对的生化差异转化为序数编码的k-mers词汇,再通过Doc2Vec模型生成交互嵌入用于下游预测任务(图1c)。相较于传统蛋白质语言模型(pLMs)的残基聚合方法,SWING能直接捕获相互作用残基的关键功能特征,在预测pMHC相互作用和错义突变效应等任务中表现出优异的零样本学习、迁移学习和泛化能力(图1d)。这些结果表明SWING通过学习相互作用残基的生化基序语义特征,为蛋白质相互作用预测提供了新的解决方案。
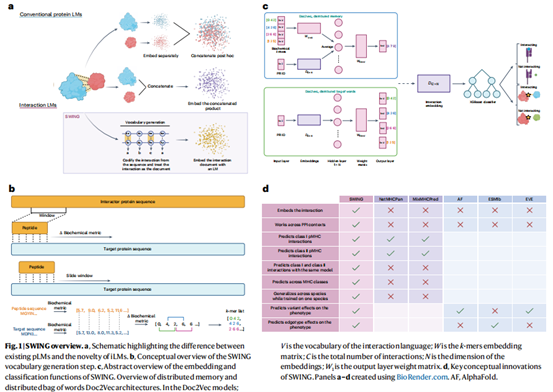
图1 | SWING概述
SWING通过创新的滑动窗口方法(图2a)将MHC功能子序列与肽段特征转化为生化语言,成功预测了pMHC-I和pMHC-II的相互作用。在交叉验证测试中(图2b-e),SWING对pMHC-I结合的预测AUC达0.72(P<0.001),并能泛化至未见等位基因(图2f-g,AUC=0.63-0.84)。对于pMHC-II(图2h-j),模型表现更为优异(交叉验证AUC=0.90,未见等位基因AUC=0.93-0.95)。这些结果表明,SWING能够通过学习MHC与肽段相互作用的"语言",有效克服传统方法对实验数据的依赖,为稀有和研究不足的MHC等位基因提供可靠的结合预测能力。
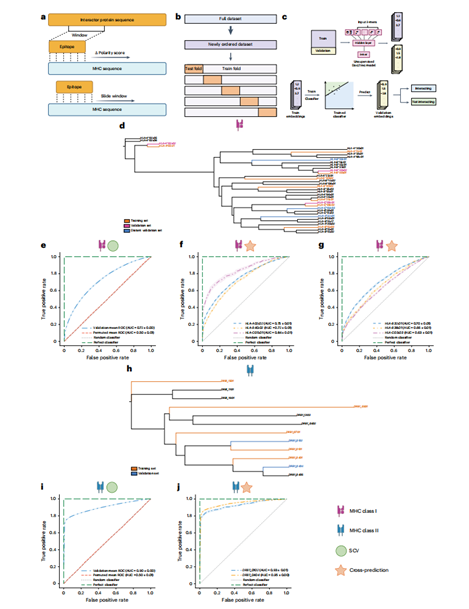
图2 | SWING预测pMHC相互作用
SWING通过系统优化训练策略和生化特征选择,证明了其模型在pMHC相互作用预测中的稳健性(图3a-i)。训练数据量测试显示,5个MHC受体已能保证模型性能(SCV-AUC=0.81),增加至12个受体未带来提升(SCV-AUC=0.72)。关键的是,SWING对生化指标的选择具有鲁棒性,无论是基于极性还是疏水性(图3a-c),I类(AUC=0.72)和II类(AUC=0.88)模型均保持优异表现。在序列长度分析中(图3d-f),使用MHC前206个氨基酸与全长序列效果相当(I类AUC=0.69,II类AUC=0.90)。特别值得注意的是,SWING能自动识别9-AA核心基序的关键作用(图3g-i),当肽段短于9个AA时性能显著下降,这一发现证实了模型捕获的是真实的生物学相互作用特征,而非人工设定的模式。
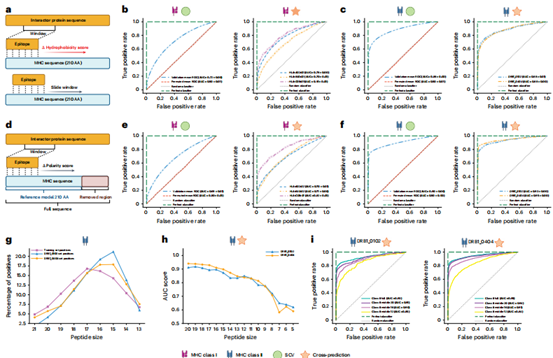
图3 | SWING捕捉pMHC生物学特性
SWING通过其独特的交互语言模型架构,在跨类别和跨物种的pMHC相互作用预测中展现出卓越的零样本学习能力(图4a-i)。其I类模型能可靠预测II类pMHC相互作用(图4b,AUC=0.74-0.77),显著优于NetMHCpan I类模型(图4c),而联合训练模型可同时预测I/II类互作(图4d-f,I类AUC=0.70-0.83,II类AUC=0.88-0.93)。更值得注意的是,未经重新训练的人类SWING模型可直接预测进化距离显著的小鼠MHC-II互作(图4g-i,AUC=0.85-0.88),这一跨物种迁移能力是现有pMHC预测工具所不具备的。这些结果表明SWING成功捕获了MHC抗原结合的保守生物学机制,为解决II类pMHC数据稀缺问题提供了创新方案。
SWING在免疫疾病相关的pMHC相互作用预测中展现出卓越性能(图4j-l)。针对狼疮肾炎相关的小鼠H-2-IEk等位基因,SWING模型(I类/II类/混合)对训练集外等位基因的预测显著优于MixMHC2pred和NetMHCIIpan,且不受肽段长度限制(13-21AA)。在1型糖尿病相关的小鼠H-2-IAg7等位基因(零样本预测)中,SWING以最高召回率准确识别结合肽段(图4k)。特别值得注意的是,SWING是唯一能有效预测IEDB全范围肽段长度(22-36AA)的方法(扩展数据图2c,AUC=0.8-0.91),在三种稀有人类自身免疫病相关II类等位基因预测中表现优异。这些结果表明SWING在预测新等位基因和非标准长度肽段相互作用方面具有独特优势。
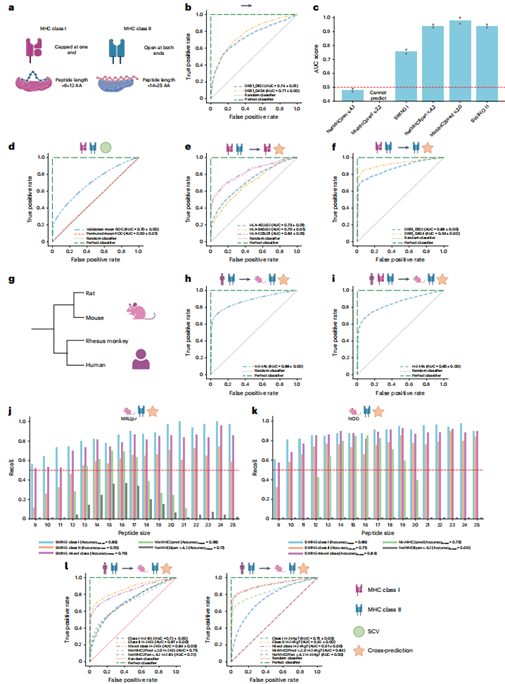
图4 | SWING零样本预测性能
SWING在预测变异对蛋白质相互作用(PPI)的特异性破坏方面展现出独特优势(图5d)。该模型能准确预测孟德尔疾病变异(图5e,AUC=0.87)、群体常见/稀有变异(图5f,AUC=0.80)以及独立于变异背景的相互作用破坏(图5g,AUC=0.81)。通过序列相似性簇分析(图5h,i)证实其稳健性(AUC=0.71),且SWING评分与等位基因频率显著相关(图5k)。典型案例分析显示(图5l),SWING能识别界面外变异(如PRKCH rs2230500和HPX rs12117)引起的罕见相互作用破坏,而AlphaMissense误判为良性。这些结果表明SWING在预测变异-表型关联方面优于传统VEP方法(图5j)。
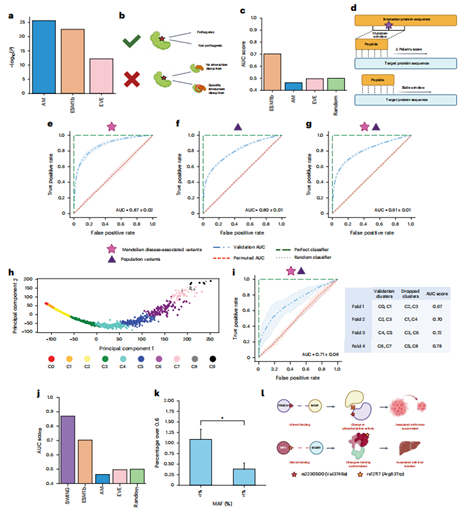
图5 | SWING预测相互作用破坏效应
SWING-iLM在蛋白质相互作用预测任务中展现出显著优势(图6a)。在突变引起的相互作用破坏预测中(图6b),SWING-iLM(AUC=0.81)优于CNN-iLM(0.74)、BERT-iLM(0.71)等对比模型,仅ESum-pLM在错义突变预测中略优(AUC=0.85)。在pMHC结合预测任务中(图6c-e),SWING-iLM对II类pMHC预测表现最佳(AUC=0.90),而XGB-iLE在I类预测中表现最优(AUC=0.89-0.96)。特别值得注意的是,SWING-iLM是唯一能实现跨类别(图6f,g,AUC=0.74-0.77)和跨物种(图6h,i,AUC=0.81-0.90)pMHC相互作用预测的模型,其他方法在这些挑战性任务中表现随机。这些结果表明,SWING-iLM在捕获蛋白质相互作用特征方面具有独特优势,尤其在跨类别和跨物种预测场景中展现出不可替代的性能。
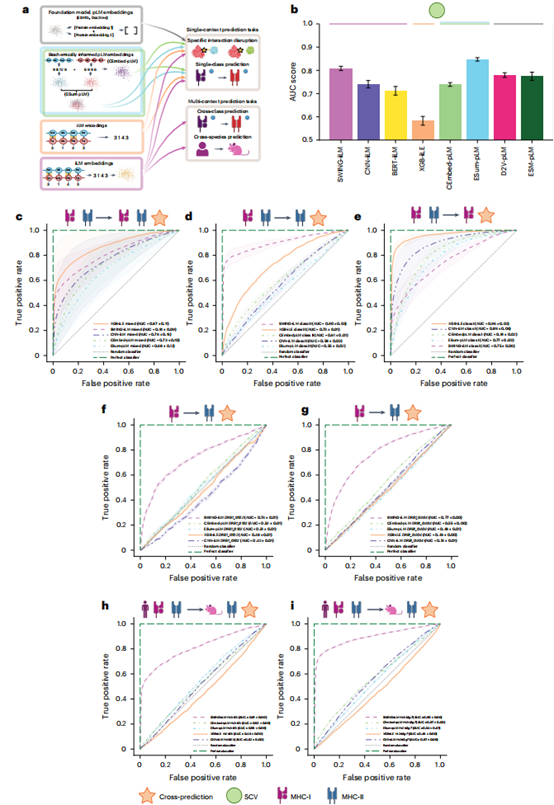
图6 | SWING与相互作用建模方法的对比
SWING作为一种创新的交互语言模型(iLM),通过将蛋白质相互作用转化为生化语言并利用Doc2Vec生成嵌入表征(图1a),成功克服了传统pLM在捕捉相互作用细微特征方面的局限。该方法仅需序列信息即可精准预测pMHC相互作用(图2-4)和变异效应(图5),并展现出跨类别(I/II类MHC)和跨物种(人类-小鼠)的迁移能力(图6)。虽然当前主要应用于蛋白质-肽段相互作用,但其框架可扩展至多分子相互作用(如TCR-pMHC复合物)及DNA/RNA-蛋白质互作等场景。值得注意的是,SWING并非通用生物基础模型,而是需要针对特定生物场景定制训练,这源于生物网络的固有残缺性和环境依赖性。相较于D-SCRIPT等全长蛋白质预测方法,SWING在肽段相关相互作用预测中具有独特优势,其学习到的交互语言特征使其成为数据稀缺场景下的有力工









