2025.09.22~2025.09.28
CNS刊登文章
01
Nature
2025/09/24
“Design of facilitated dissociation enables timing of cytokine signalling”
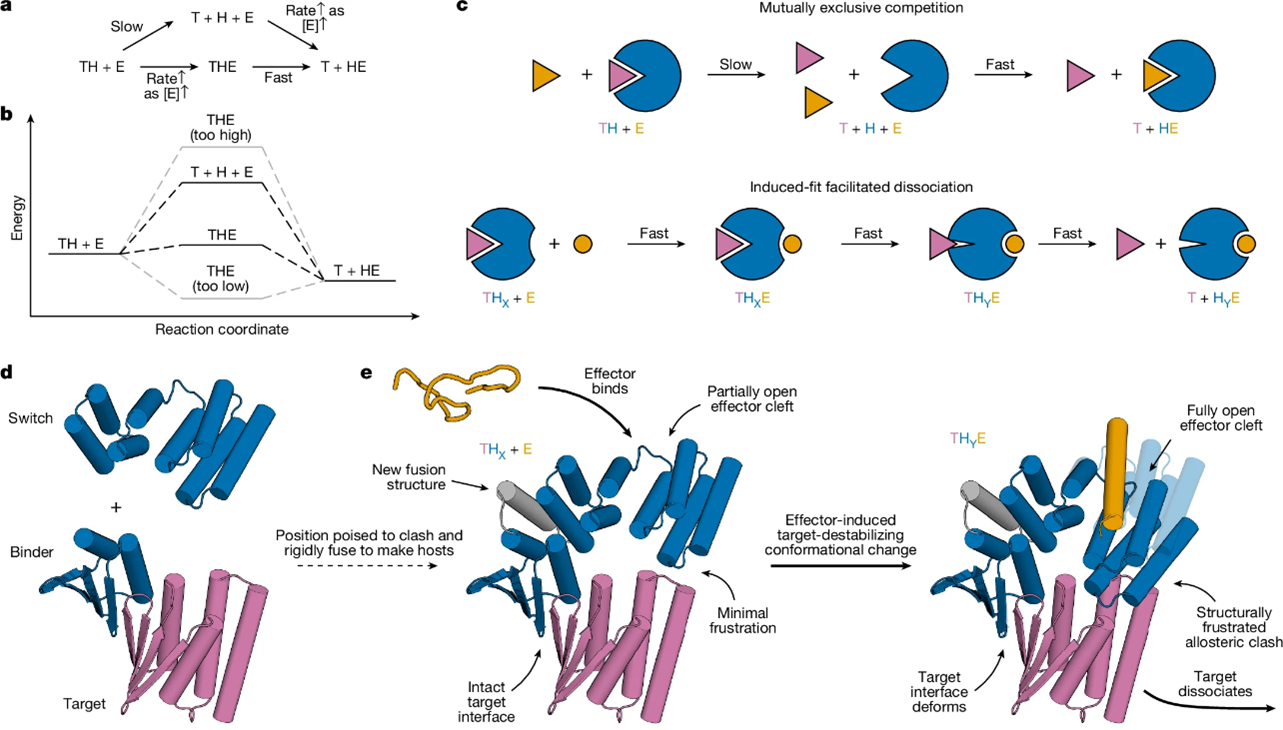
蛋白质设计主要集中在设计基态,确保它们能量足够低,从而高度占据。若要设计体系的动力学和动态特性,则需要进一步设计在从一个低能态过渡到另一个低能态过程中所经历的激发态。这是一项极具挑战性的任务,因为这些激发态必须足够受限以保持低占据,但又不能受限到完全不被占据;同时,以往的蛋白质设计方法大多专注于生成近乎理想的结构。
来自美国华盛顿大学的David Baker和Adam J. Broerman以及奥地利科学技术研究所的Florian Praetorius团队合作提出了一种通用策略:通过诱导契合的动力冲程,生成结构受挫且受限的激发态,从而别构性地驱动蛋白质复合物的解离。X射线晶体学、双电子–电子共振(DEER)谱和动力学结合测量表明,激发态的引入使得设计的效应子可诱导解离速率最高增加至 5700 倍。研究者通过设计快速生物传感器、动力学可控电路和能够在数秒内与受体解离的细胞因子模拟物,展示了该方法的威力,从而能够解析白细胞介素-2 信号传导的时间动态过程。
02
Science
2025/09/25
“Predicting protein-protein interactions in the human proteome”
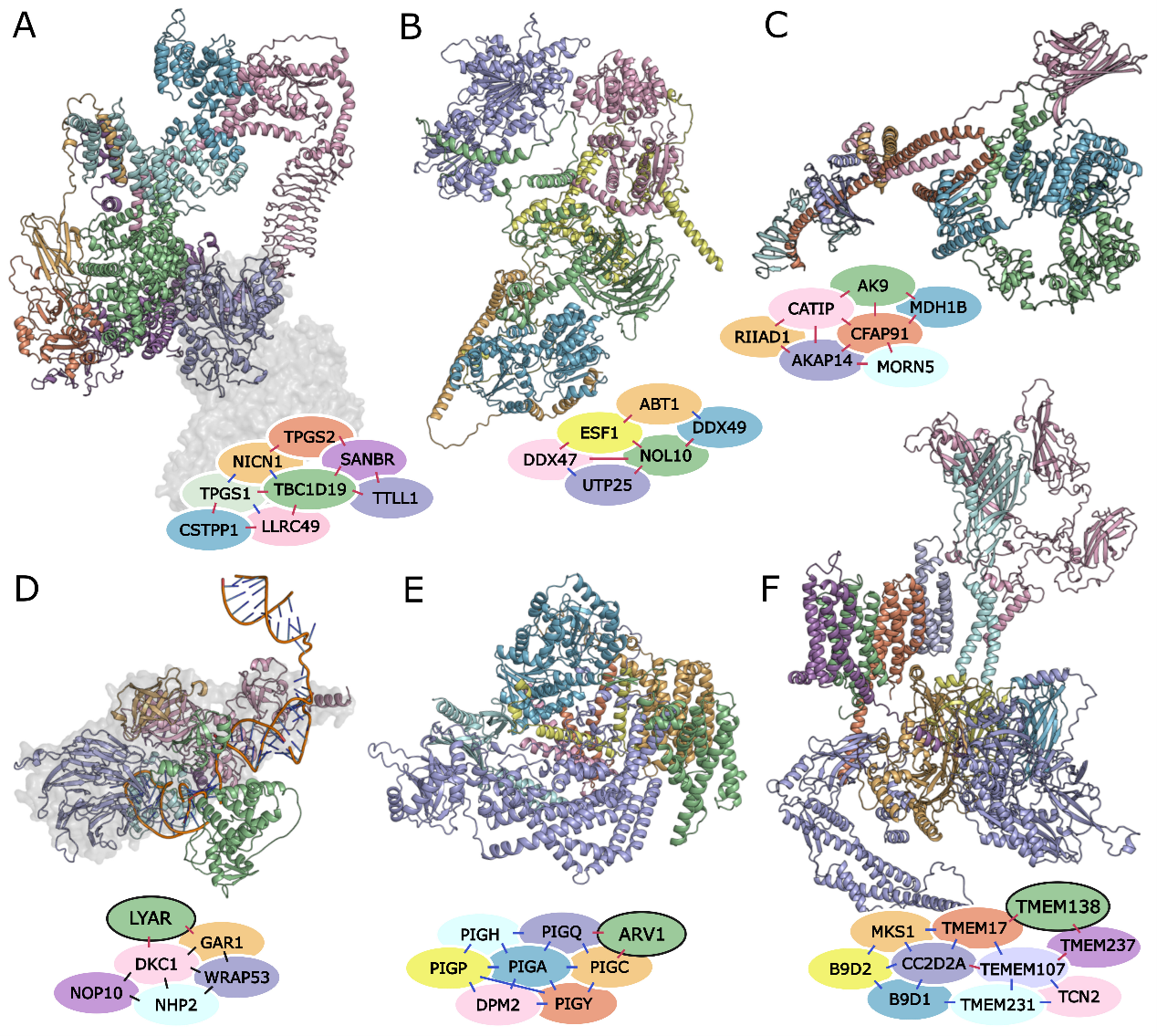
蛋白质–蛋白质相互作用(PPI)对生物学功能至关重要。共进化分析和基于深度学习(DL)的蛋白质结构预测已经使在细菌和酵母中实现全面的 PPI 鉴定成为可能,但这些方法在更复杂的人类蛋白质组中成功有限。
来自美国德克萨斯大学西南医学中心的丛倩团队通过两方面克服了这一挑战:其一是利用 30 PB 未组装基因组数据构建的多序列比对,使共进化信号加深了 7 倍;其二是开发了一种新的深度学习网络,该网络在包含 2 亿个预测蛋白质结构的结构域–结构域相互作用增强数据集上进行训练。研究者系统性地筛查了 2 亿个人类蛋白质对,预测出 17,849 个相互作用,预期准确率为 90%,其中有 3,631 个相互作用在先前的实验筛查中未被发现。这些预测相互作用的三维模型为蛋白质功能及人类疾病机制提供了大量新的假设。
03
Cell
2025/09/22
“Time-resolved fluorescent proteins expand fluorescent microscopy in temporal and spectral domains”
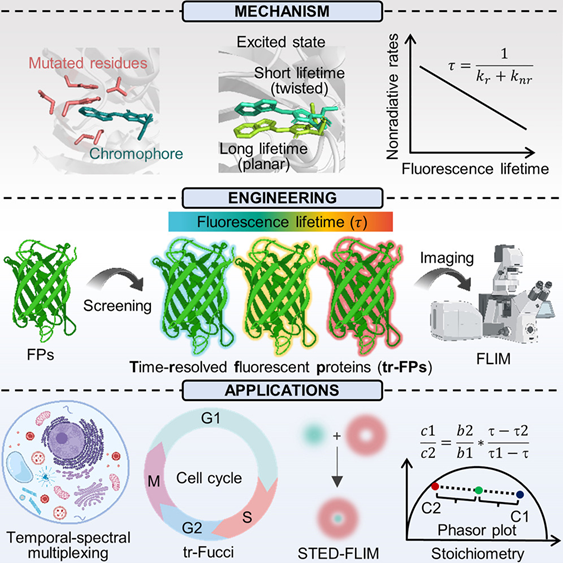
荧光显微技术已被广泛应用于生命科学研究。虽然荧光强度作为稳态信号被普遍使用,但基于荧光寿命的时间分辨(tr)信号仍然研究不足。
来自西湖大学张鑫团队提出了一类具有合理控制寿命的时间分辨荧光蛋白(tr-FPs)。通过一种在不改变荧光蛋白光谱的前提下调控寿命的策略,研究者开发出一系列覆盖可见光谱并具有广泛寿命范围的 tr-FPs。这些 tr-FPs 被应用于时域-光谱分辨显微成像,能够在活细胞中同时成像 9 种不同的蛋白质,并将多种活性与细胞周期相关联。此外,tr-FPs 还能实现多路超分辨显微成像,利用生命周期信号同时观察 4 种蛋白质,并被用于定量测定细胞蛋白质的化学计量比。本研究引入了 tr-FPs 的概念与开发,作为一种变革性的工具集,为将系统复杂性与定量精确性相结合融入生物学研究提供了新机遇。
2025.09.22~2025.09.28
子刊刊登文章
01
Cell Research
9.29
“Molecular basis of vitamin K-dependent protein γ-glutamyl carboxylation”
02
Molecular Cell
本周无
03
Nature Structural & Molecular Biology
9.22
9.25
2.“Cryo-EM structures reveal the molecular mechanism of SUMO E1–E2 thioester transfer”
9.29
3.“Molecular basis for the activation of outer dynein arms in cilia”
04
Nature Communications
9.22
1.“Repeatability of protein structural evolution following convergent gene fusions”
9.24
2.“Structural remodeling of target-SNARE protein complexes by NSF enables synaptic transmission”
9.24
3.“Periplasmic protein quality control at atomic level in live cells”
9.25
9.25
9.25
9.25
9.26
9.26
9.26
9.26
11.“A domain-swapped CaMKII conformation facilitates linker-mediated allosteric regulation”
9.26
12.“Structural basis of antiphage defence by an ATPase-associated reverse transcriptase”
9.29
9.29
9.29
15.“Molecular mechanisms of SLC30A10-mediated manganese transport”
9.29
16.“Molecular mechanisms of CBASS phospholipase effector CapV mediated membrane disruption”
05
Science Advances
9.24
9.24
2.“Structural basis of Pseudomonas biofilm-forming functional amyloid FapC formation”
9.24
3.“Cryo-EM snapshots of NMDA receptor activation illuminate sequential rearrangements”
9.24









