2025.12.01~2025.12.07
CNS刊登文章
01
Nature
2025/12/03
1.“Computational enzyme design by catalytic motif scaffolding”
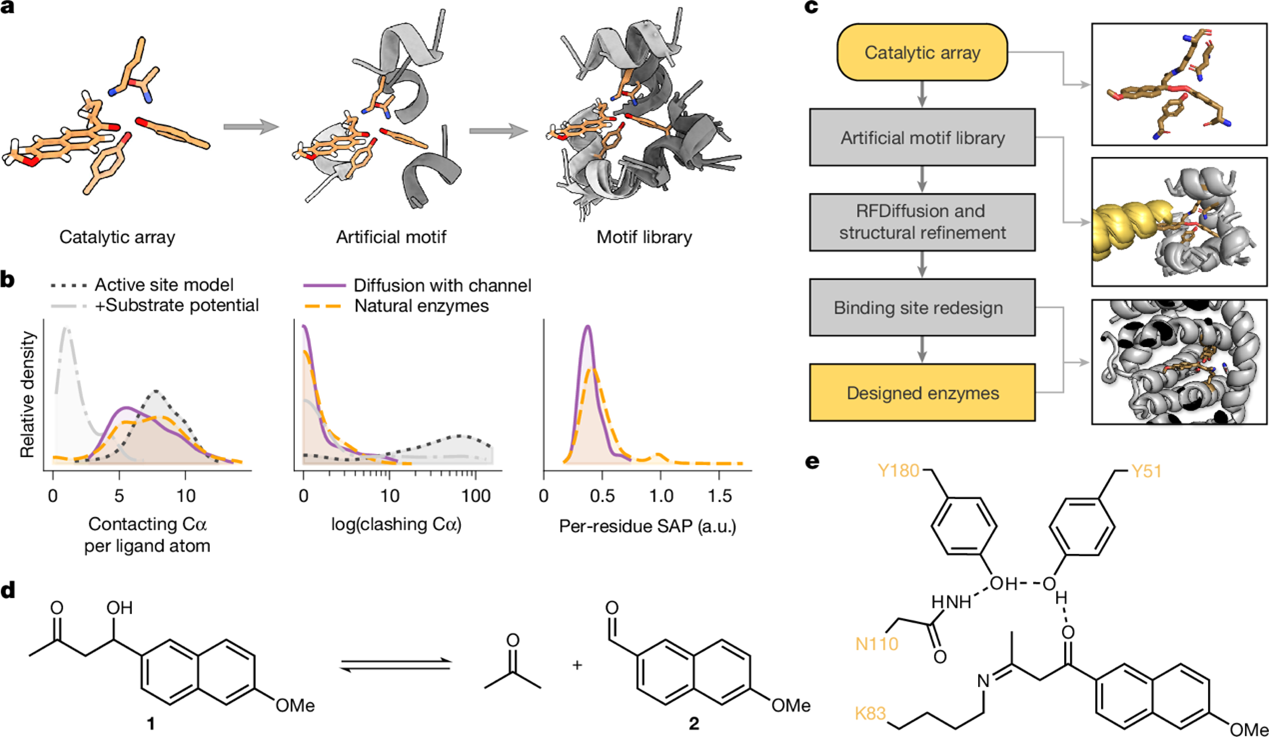
酶因其高度精确的选择性、高效性以及温和的反应条件,而在工业和医学中被广泛用作生物催化剂。通过定制设计酶,可以构建能够执行天然反应之外的特定反应的生物催化剂。然而,现有的设计方法需要测试大量的设计,并且大多数得到的 de novo 酶催化活性较低。因此,这些方法往往需要昂贵的实验优化和高通量筛选,才能在工业上具有可行性。
来自奥地利格拉茨工业大学的Gustav Oberdorfer课题组提出了 rotamer inverted fragment finder–diffusion(Riff-Diff),一种将机器学习与原子级建模结合的策略,用于在全新设计的蛋白质中构建催化基团组合。研究者通过设计两类在机理上截然不同的化学反应的酶——逆醛缩反应(retro-aldol)和 Morita–Baylis–Hillman(MBH)反应——展示了 Riff-Diff 的广泛适用性。研究者表明,在这两种情形中,都能够生成催化剂,其活性可与经体外进化优化后的酶媲美,并表现出极高的立体选择性。六个设计的高分辨率结构显示,其活性位点的构建达到了近乎原子级的精确度。该设计策略原则上可应用于任何具有催化能力的氨基酸阵列。这些发现为 de novo 蛋白催化剂在合成中的实际应用奠定了基础,并揭示了蛋白质设计与酶催化的基本原理。
2025/12/03
2.“Computational design of metallohydrolases”
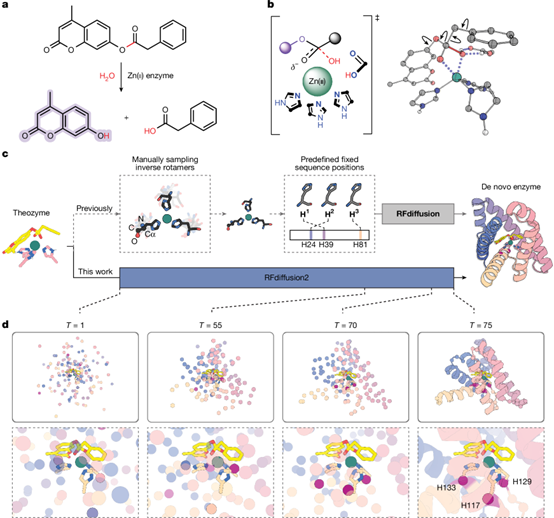
从头设计酶(de novo enzyme design)的目标是构建能够包含理想活性位点的蛋白质,使催化残基围绕并稳定目标化学反应的过渡态。生成式人工智能方法 RFdiffusion能够解决这一问题,但它需要为每个催化残基同时指定序列位置和主链坐标,从而限制了采样空间。
来自美国华盛顿大学David Baker和Indrek Kalvet以及瑞士苏黎世联邦理工学院的Donald Hilvert课题组合作提出 RFdiffusion2,一种消除了上述限制的新方法,并利用其基于量子化学推导的活性位点几何结构,设计了锌金属水解酶(zinc metallohydrolases)。在第一轮的 96 个实验测试的设计中,活性最高的变体的催化效率(kcat/KM)达 16,000 M⁻¹ s⁻¹,比之前设计的金属水解酶高出数个数量级。第二轮的 96 个设计进一步获得了 3 个高活性酶,kcat/KM 最高可达 53,000 M⁻¹ s⁻¹,其催化速率常数(kcat)最高达到 1.5 s⁻¹。
这四个最高活性的设计模型彼此之间以及与已知结构均明显不同,而最活跃酶的晶体结构与其设计模型高度一致,表明该设计方法具有很高的准确性。PLACER和 Chai-1预测这些高活性酶具有预组织化的活性位点,能够有效定位底物,使其可被由金属离子活化的水分子进行亲核攻击。能够直接从计算机中设计出无需实验优化的高活性酶,意味着新一代高效、可定制的催化剂将成为可能。
02
Science
2025/12/04
1.“Multiscale structure of chromatin condensates explains phase separation and material properties”
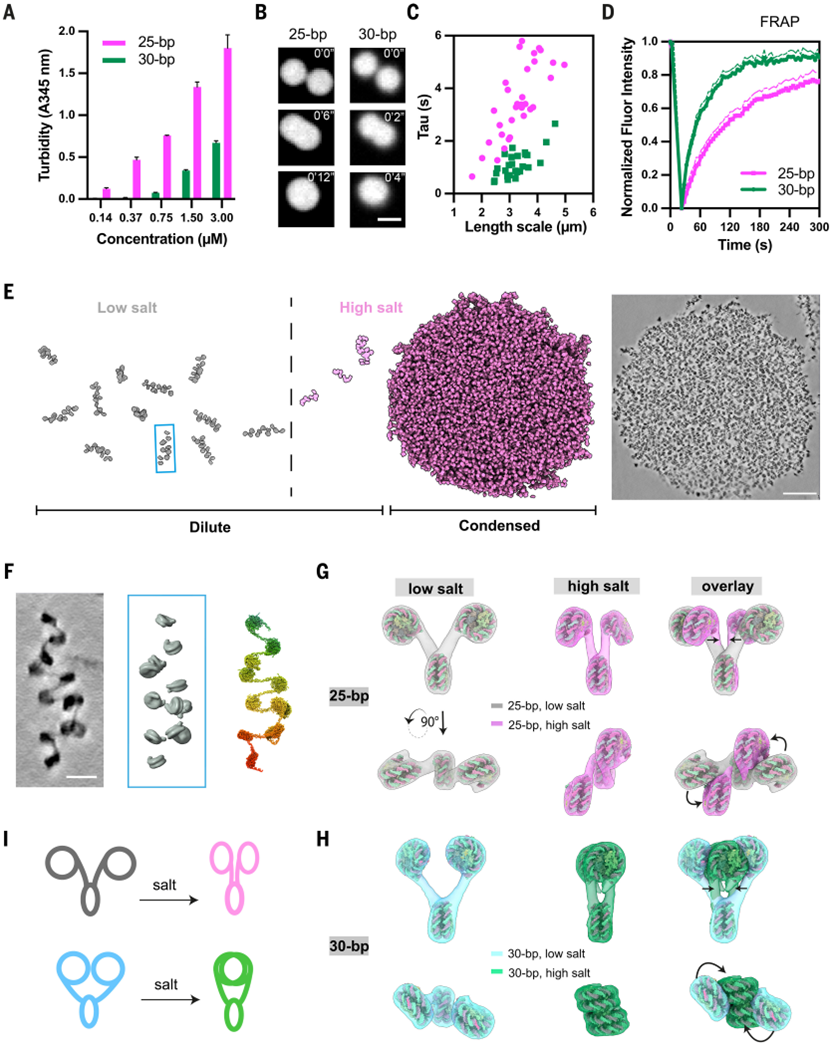
生物大分子凝聚体内部的分子结构及其相互作用网络长期以来尚不清晰。通过冷冻电子断层扫描(cryo–electron tomography)结合分子动力学模拟,研究者解析了相分离染色质凝聚体的结构,其尺度跨越从单个氨基酸到整体网络结构。
来自美国德克萨斯大学西南医学中心的Michael K. Rosen和海洋生物实验室染色质协作组的Rosana Collepardo-Guevara课题组合作研究发现,核小体间 DNA linker 的长度决定了核小体的排列方式及组蛋白尾部的相互作用,从而塑造了单个染色质分子在凝聚体内外的结构。这种结构上的调节作用决定了分子内与分子间相互作用的平衡,而这种平衡又控制着染色质凝聚体的分子网络、热力学稳定性与材料特性。哺乳动物细胞核中存在致密的核小体簇,其非随机组织方式与体外重构的凝聚体高度一致。本研究揭示了单个染色质分子的结构如何决定染色质凝聚体的物理性质及细胞内染色质的整体组织方式。
03
Cell
2025/12/01
1.“An archaeal transcription factor bridges prokaryotic and eukaryotic regulatory paradigms”
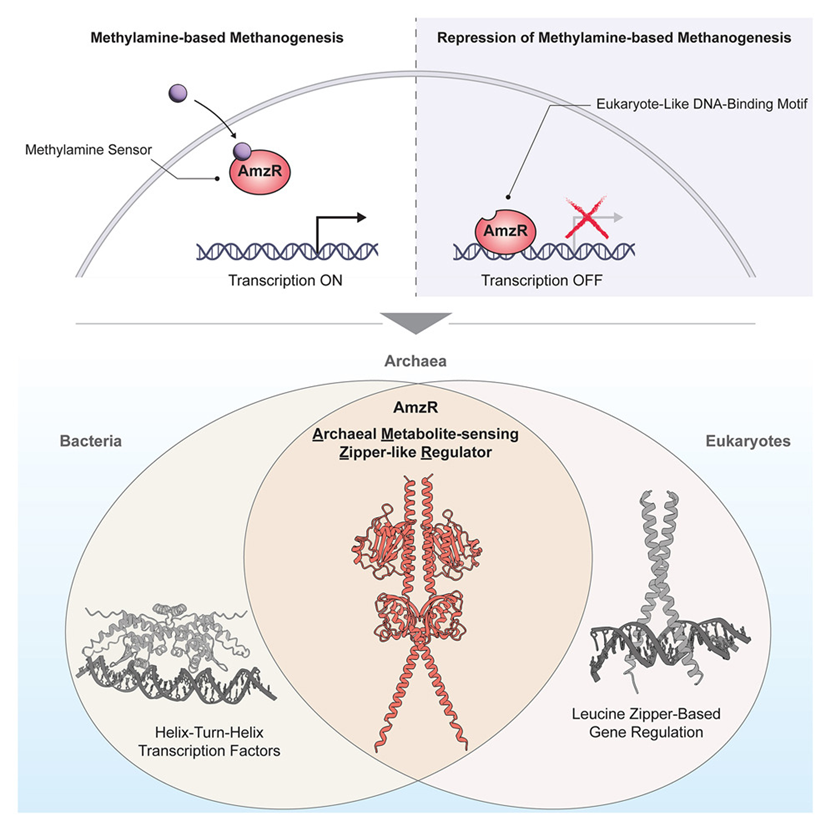
古菌的转录机制融合了真核生物和原核生物的特征:一种类似 RNA 聚合酶 II(RNAPII)的聚合酶负责转录环状染色体上组织的基因,而这些细胞本身并不具有细胞核。因此,与其他生命界相比,古菌基因组缺乏常见的转录调控因子。
来自美国加利福尼亚大学伯克利分校的Fernando Medina Ferrer和Dipti D. Nayak课题组合作描述了一个隐蔽的、古菌特有的配体结合型转录调控因子(TF)家族的发现,该家族被命名为 AmzR(archaeal metabolite-sensing zipper-like regulators,古菌代谢物感受类拉链样调控因子)。研究者通过基于进化的遗传筛选方法鉴定出 AmzR,并证明它是产甲烷古菌 Methanosarcina acetivorans 中调控甲胺类底物产甲烷生长的抑制因子。AmzR 通过成寡聚体的方式结合其靶启动子,其结合依赖成对的碱性 α 螺旋结构,这与真核生物中的亮氨酸拉链(leucine zipper)类似。AmzR 还能够结合甲胺类小分子,这种结合会降低其与 DNA 的亲和力,使其作为一种原核生物中常见的一组分系统发挥功能,同时却具有类似真核生物的 DNA 结合结构域。AmzR 家族的转录因子在古菌中广泛存在,拓展了位于原核—真核交界处的生物创新范畴。
2025/12/02
伞形毒素的独特结构使其能够采用两级分子风险对冲策略,实现细菌间的拮抗作用
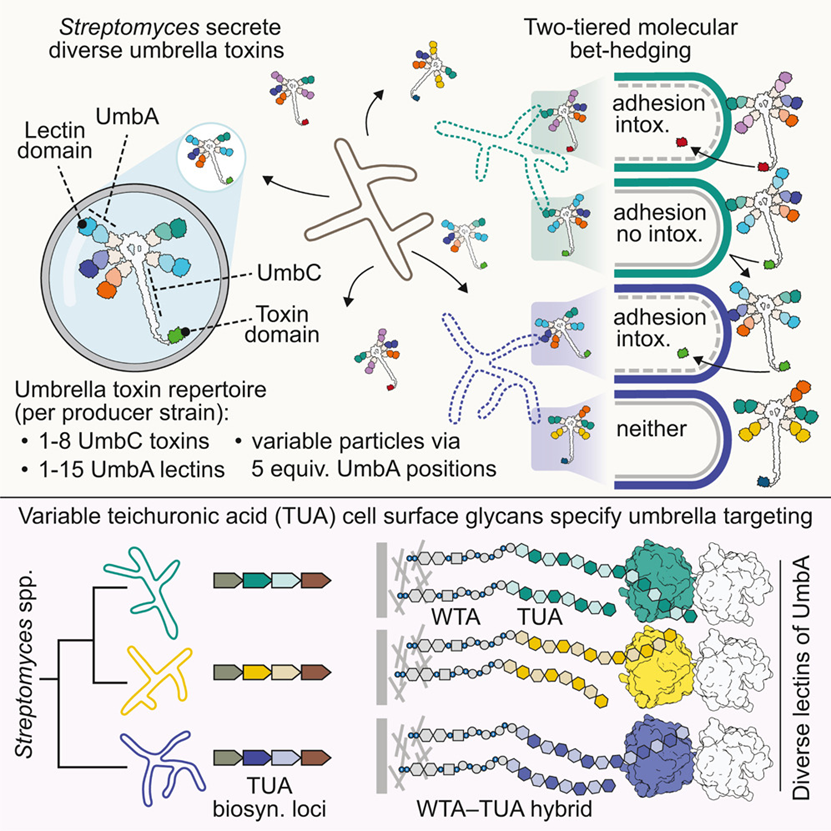
细菌生活在竞争激烈且迅速变化的环境中,在其中未来威胁的性质往往难以预测。链霉菌 Streptomyces coelicolor 会产生三种“伞状颗粒(umbrella particles)”抗菌因子,这些颗粒包含不同的多态性毒素结构域,以及一组高度多样化、但部分重叠的六种凝集素(lectins)。
来自美国华盛顿大学Joseph D. Mougous课题组表明这些伞状颗粒的高度特异性来源于凝集素介导的对特定物种的结合,其靶点是此前未被描述的高度可变的表面糖缀合物(surface glycoconjugates)。研究者利用单颗粒冷冻电镜(cryo-EM)解析了其中一种凝集素与其寡糖底物的复合物结构,从而揭示了其靶向机制的分子基础——即通过对多个糖链特征的协调识别来实现结合。对多种靶物种的生化和遗传研究,结合凝集素交换实验(lectin-swapping experiments),支持了这样一个模型:S. coelicolor 通过在凝集素组成和毒素多态性两个层面上对伞状毒素进行多样化,是一种独特的、双层级的风险分散(bet-hedging)策略。生物信息学分析进一步支持这一观点,即这种非典型结构的伞状毒素为链霉菌提供了一种可泛化的策略,使其能够对抗难以预测的多种竞争者。
2025/12/04
3.“A fin-loop-like structure in GPX4 underlies neuroprotection from ferroptosis”
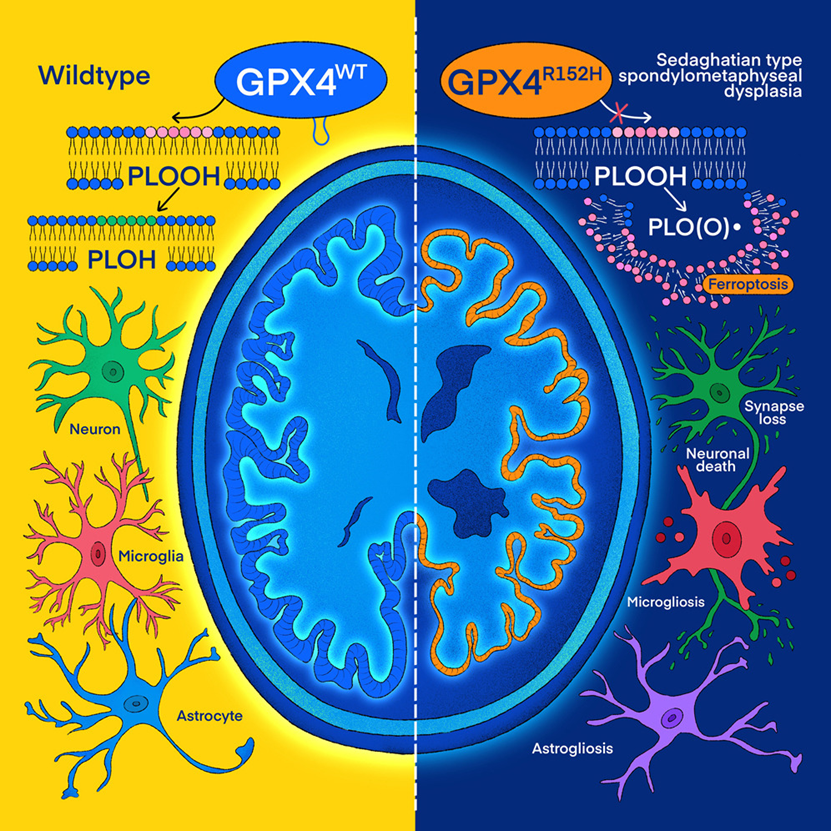
铁死亡是一种由细胞膜磷脂的失控过氧化驱动的独特细胞死亡方式,它不同于其他类型的细胞死亡,因为铁死亡并没有明确的起始信号,同时受到内源性抗氧化系统的持续监控。作为铁死亡的核心守护者,谷胱甘肽过氧化物酶 4(GPX4)负责保护细胞膜免受过氧化损伤,但其发挥保护作用的具体机制长期以来仍不清楚。
来自德国代谢和细胞死亡研究所Marcus Conrad课题组通过对 GPX4 中一个导致早发性神经退行性疾病的错义突变(p.R152H)进行结构和功能分析,发现该突变显著削弱了 GPX4 的膜锚定能力,而其催化活性却基本保持完好。在小鼠中,时空性删除 Gpx4 或使神经元特异性表达 GPX4R152H 均会造成大脑皮层与小脑神经元的退化,并伴随逐渐加重的神经炎症。来自患者的诱导多能干细胞(iPSC)分化得到的皮层神经元和前脑类器官也表现出更高的铁死亡敏感性,重现了关键病理特征,并对铁死亡抑制剂产生明显反应。神经蛋白质组学分析进一步显示,受损脑组织中出现了类似阿尔茨海默病的分子特征。这些结果共同表明,GPX4 正确的膜锚定对于其保护功能至关重要,也确立了铁死亡在神经退行性变中的核心驱动作用,从而为将铁死亡作为治疗神经退行性疾病的潜在靶点提供了理论依据。
2025.12.01~2025.12.07
子刊刊登文章
01
Cell Research
本周无
02
Molecular Cell
本周无
03
Nature Structural & Molecular Biology
12.01
1.“DNA topoisomerase I acts as supercoiling sensor for bacterial transcription elongation”
12.03
12.05
3.“UBA6 specificity for ubiquitin E2 conjugating enzymes reveals a priority mechanism of BIRC6”
04
Nature Communications
12.01
1.“Molecular mechanism of PINK1 regulation by the Hsp90 machinery”
12.01
12.01
3.“RAD51 D-loop structures reveal the mechanism of eukaryotic RAD51-mediated strand exchange”
12.01
4.“Rapid estimation of protein folding pathways from sequence alone using AlphaFold2”
12.01
5.“Molecular basis of prostaglandin E2 reuptake by organic anion transporter PGT”
12.02
12.02
7.“Understanding interspecies drug response variations between human and rodent P2X7 receptors”
12.02
8.“Identification and characterization of binders to a cryptic and functional pocket in KRAS”
12.02
9.“Structural mechanism of the Retron-Eco7 anti-phage defense system”
12.02
12.02
12.03
12.“Molecular basis for multidrug efflux by an anaerobic-associated RND transporter”
12.04
13.“Molecular insights into species-specific ACE2 recognition of coronavirus HKU5”
12.04
14.“Discovery of an allosteric binding site for anthraquinones at the human P2X4 receptor”
12.04
15.“The Human T-cell Leukemia Virus capsid protein is a potential drug target”
12.04
12.04
17.“KY216-tubulin complex captures VASH2 to inhibit NSCLC metastasis”
12.04
18.“Structural basis of specific lysine transport by Pseudomonas aeruginosa permease LysP”
12.04
19. “Benchmarking all-atom biomolecular structure prediction with FoldBench”
12.05
20.“Structural mechanism of mRNA decoding by mammalian GTPase GTPBP1”哺乳动物
12.05
12.05
22.“Lipid dependence of connexin-32 gap junction channel conformations”
12.06
12.06
24.“Ion channel inhibition by targeted recruitment of NEDD4-2 with divalent nanobodies”
12.06
25.“Temporally resolved and interpretable machine learning model of GPCR conformational transition”
12.06
26.“Structural insights into VLDLR recognition by western equine encephalitis virus”
12.07
27.“Architecture and mechanism of a dual-enzyme retron system in prokaryotic immunity”
12.07
28.“Structural-guided identification of two modulators of β−1,3-glucan synthase FKS1”
05
Science Advances
12.03
12.03
12.03
12.03









