2025.12.15~2025.12.21
CNS刊登文章
01
Nature
2025/12/22
“Non-equilibrium snapshots of ligand efficacy at the μ-opioid receptor”
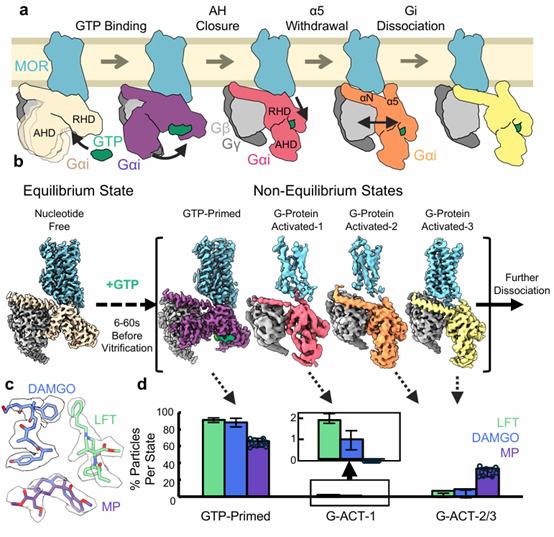
不同配体作用于同一 G 蛋白偶联受体(GPCR)时,会在不同程度上激活细胞内信号通路,但驱动这些差异的分子机制仍不清楚。
来自美国斯坦福大学医学院的Georgios Skiniotis课题组假设,这种信号效能的差异可以在非平衡条件下的中间态中通过结构方式被捕捉。基于这一假设,研究者采用时间分辨(time-resolved, TR)冷冻电镜(cryo-EM)方法,观察在三种分别表现为部分激动剂、完全激动剂或超激动剂的配体结合状态下,μ-阿片受体(MOR)诱导 GTP 触发的 Gαiβγ 异源三聚体激活过程。研究者解析了沿着 G 蛋白激活通路分布的一系列构象状态,其中包括一种此前未被观察到的中间态,能够直接可视化受体动力学如何随结合配体而变化。结果显示,不同配体在状态占据比例和构象稳定性上存在显著差异,且配体效能越高,受体跨膜螺旋 TM5 和 TM6 的构象动态性越强。此外,研究者还鉴定了 Gi 与 Gs 在 GTP 诱导激活过程中的关键机制差异,这些差异可能是二者激活动力学不同的根本原因。结合分子动力学(MD)模拟和单分子荧光实验的验证,这些结果共同描绘了不同效能配体条件下 GPCR–G 蛋白相互作用的动态结构图景,并提示部分激动剂可能在 G 蛋白激活过程中形成一种“动力学陷阱”。
02
Science
2025/12/18
“Conformational landscape adaptations enable processive phosphorylation by Src family kinases”
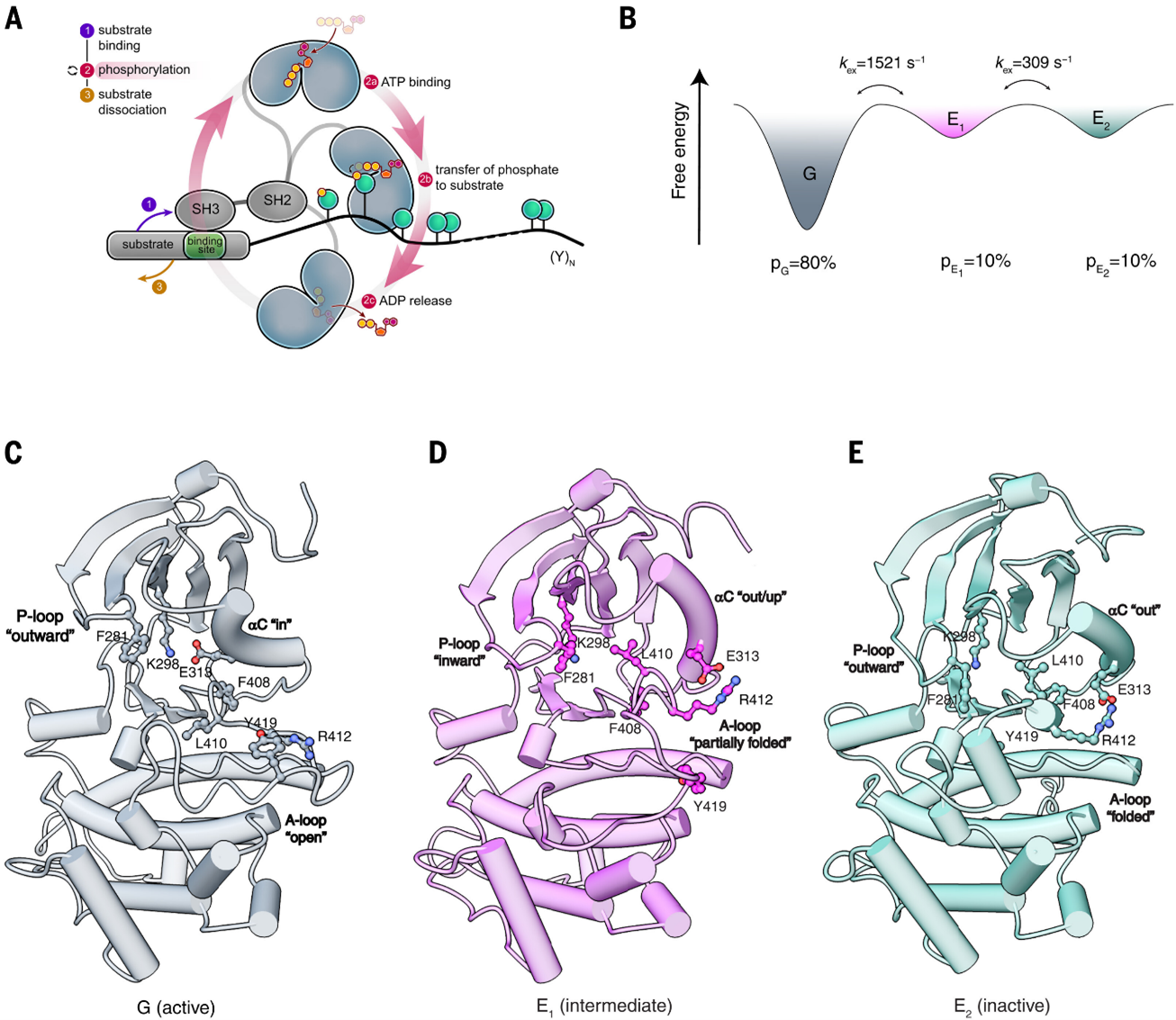
激酶介导的过程性磷酸化(processive phosphorylation)使信号枢纽蛋白能够在短时间内完成多位点修饰,从而在时间敏感的细胞事件中整合多种信号。要实现过程性磷酸化,底物在解离之前必须经历多个催化循环,因此快速的催化周转率至关重要。Src 家族激酶能够对多位点底物进行过程性磷酸化。
通过核磁共振(NMR)谱学研究,来自圣犹达儿童研究医院Charalampos G. Kalodimos课题组在 Src 的构象集合中鉴定到一种瞬态中间态,其构象位置介于活化态与失活态之间。该中间态有助于在 ATP 水解后快速释放二磷酸腺苷(ADP),从而保证高效的催化周转。去除这一中间态会使 Src、Lck 和 Hck 的过程性磷酸化能力丧失,并导致其功能受损。这些研究结果表明,Src 家族激酶的构象集合在进化过程中引入了一种关键的瞬态状态,从而支撑了其对底物进行过程性磷酸化的能力。
03
Cell
本周无
2025.12.15~2025.12.21
子刊刊登文章
01
Cell Research
本周无
02
Molecular Cell
12.19
“Toward community-driven visual proteomics with large-scale cryo-electron tomography of Chlamydomonas reinhardtii”
03
Nature Structural & Molecular Biology
本周无
04
Nature Communications
12.15
12.15
2.“Structure and mechanism of the broad spectrum CRISPR-associated ring nuclease Crn4”
12.15
3.“Structural basis for genome-wide site-specific DNA recognition by Nuclear Factor IA”
12.16
4.“The resting and ligand-bound states of the membrane-embedded human T-cell receptor–CD3 complex”
12.16
12.16
6.“Molecular insights into ago-allosteric modulation at cysteinyl leukotriene receptor 2”
12.16
7.“Structural basis for ACT1 oligomerization induced by IL-17 receptor hetero-tetramer”
12.16
12.16
9.“Calcium-sensitive synaptotagmin 11-lipid interaction modulates exo-endocytosis”
12.16
10.“Structure of ATTRv-F64S fibrils isolated from skin tissue of a living patient”
12.16
11.“Molecular basis of XPF-ERCC1 targeting to SLX4-dependent DNA repair pathways”
12.17
12.“Peptidoglycan recruitment by a penicillin binding protein”
12.17
12.17
12.17
15.“Structural snapshots of Pseudomonas aeruginosa LptB2FG and LptB2FGC reveal insights into lipopolysaccharide recognition and transport”
12.17
16.“Structural insights into the coordinated regulation of the SLAH family in Arabidopsis thaliana”
12.17
12.17
12.18
19.“Molecular insights into ligand recognition and signaling of OXGR1”
12.18
12.18
21.“Structural insights into cationic amino acid transport and viral receptor engagement by CAT1”
12.18
22.“Molecular mechanisms of receptor recognition and antibody neutralization of coxsackievirus A6”
12.18
23.“Structural insights into C3 convertase activity of the classical pathway of complement”
12.19
24.“Molecular basis of ApoER2-mediated Semliki Forest virus entry”
12.19
12.19
26.“Directed evolution of a TNA polymerase identifies independent paths to fidelity and catalysis”
12.20
12.20
12.21
29.“The cryo-EM structure of Photosystem I from Chromera velia with a bound superoxide dismutase heterodimer”
05
Science Advances
12.19
1.“Mechanism of cotranslational modification of histones H2A and H4 by MetAP1 and NatD”
12.19
12.19
3.“Structural basis of VLCFA chain-length determination by the KCS6-CER2 complex in plants”









